|
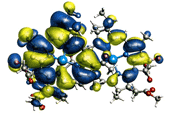
■電子状態シミュレーション1
AMOSS (Ab initio Molecular Orbital Simulation on Supercomputer)
NEC基礎研究所が開発した非経験的な分子軌道法計算のプログラム。巨大な系の並列下における高速計算、高精度計算が可能。CI( Configuration interaction:配置間相互作用)計算も開発中。(figure
1)
■電子状態シミュレーション2
DFT (Density Function Theory) / Generalized DFT
大阪大学大学院理学研究科山口兆教授、山中研究員(同客員助教授)のグループが開発した密度汎関数法の計算および一般化密度汎関数法のプログラム。通常のDFT(例えば汎用G03プログラムにあるスピン分極型DFT)では扱えないような、スピンフラストレーションのある系、スピン縮重、スピン傾斜をともなうような系に対して計算が可能。それらを基に生体化学反応、特に酸化還元反応(例えばフェレドキシン)の複雑な反応に対してシミュレーションができる。
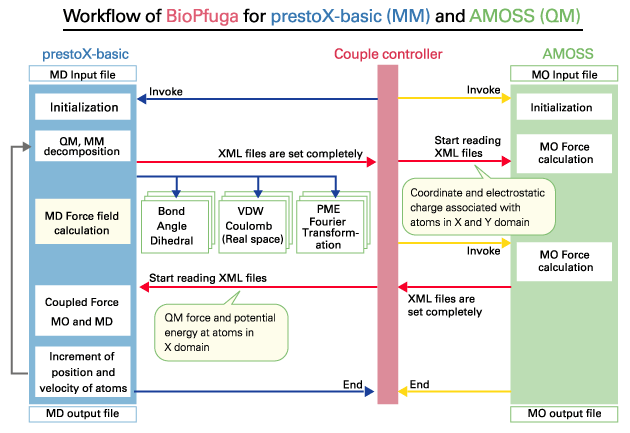
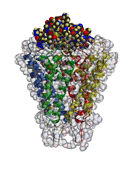
■蛋白質複合体シミュレーション
prestoX-basic
In silico drug screeningを目的に、産業技術総合研究所JBIRC、大阪大学蛋白質研究所、横浜市立大学、日立製作所、富士通で共同開発中の古典力学に基づく分子動力学計算プログラム。(figure
2)
■蛋白質フォールドシミュレーション
アミノ酸配列情報からの ab initio 蛋白質立体構造予測
神戸大学理学部高田彰二博士らの研究。国際コンテストCASPで高い評価。
■細胞生理シミュレーション
薬物によるヒト・心筋細胞のイオンチャンネルの阻害を機能的に理解するシミュレーション
大阪大学大学院医学系研究科倉智嘉久教授らの研究。
■器官・組織シミュレーション
表面筋電位発生と肺内換気分布シミュレーション
大阪大学大学院情報科学研究科赤澤堅造教授および北岡裕子博士らが開発中。筋電位信号発生シミュレーションや肺の構造機能解析による診断とシミュレーションによる治療予測
|